
能以千倍之速度找到潛在藥物分子的AI模型
科技產業資訊室 (iKnow) - 周媛韻 發表於 2022年7月26日
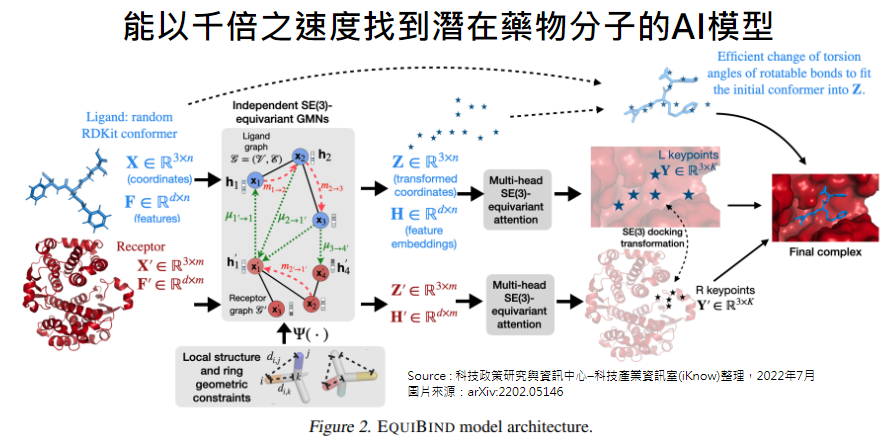
圖、能以千倍之速度找到潛在藥物分子的AI模型
麻省理工學院的研究人員開發了一種名為EquiBind的幾何深度學習模型,比現有稱為QuickVina2-W的計算分子嵌合模型(computational molecular docking models)快了1,200倍的速度,成功的將類藥物的分子及蛋白質結合。
EquiBind是EquiDock的改良版,在藥物開發之前,研究人員需要找出類藥物分子並且正確的與特定蛋白質標的綑綁及「對接」在一起。在成功與蛋白質對接後,與藥物形成配位體(ligand),可以阻止蛋白質發揮作用。若這個現象發生在細菌的必需蛋白質(essential protein)上,則可以殺死細菌進而保護人體。
然而藥物的發現(drug discovery)過程在財務或是計算上的成本都很高,每次藥物發現的過程都需要投入數十億美元,並在FDA最終批准前進行了數十年的開發及測試。並且有高達90%的藥物在進行到人體試驗階段中由於沒有任何效果或是太多副作用而失敗。製藥公司會透過提高開發成功藥物的價格來回收這些失敗的成本。
目前找出潛在藥物候選分子計算過程的最先進計算模型依賴於大量結合評分、排名及微調的候選本樣本,以獲得最佳蛋白質及配體的配適。透過EquiBind的模型,只需要一步就能直接預測精準的關鍵位置,而無需事先了解蛋白質標的,即為「盲對接(blind docking)」。
有別於大多數需要多次嘗試才能在蛋白質中的配體找到有利位置的模型,EquiBind已經內建幾何推理(geometric reasoning)功能,可以幫助模型學習分子基本的物理特性,並成功地廣泛對新的或未曾見過的資料進行更好的預測。
這個發現目前已經引起Relay Therapeutics的首席數據長使用該模型來進行現有用於肺癌、白血病及腸胃道腫瘤的藥物及蛋白質的模型測試,發現EquiBind比起過去傳統的對接方法更成功。目前研究團隊致力於了解企業專業的使用回饋,來進一步改善該模型。
該研究由Pharmaceutical Discovery and Synthesis財團、Jameel診所、DTRA Discovery of Medical Countermeasures Against New and Emerging threats計畫、DARPA Accelerated Molecular Discovery計畫、MIT-Takeda獎學金及NSF Expeditions grant Collaborative Research: Understanding the World Through Code資助,並發表在ICML研討會上。(694字;圖1)
參考資料:
Artificial intelligence model finds potential drug molecules a thousand times faster. MIT News,2022/7/12
EquiBind: Geometric Deep Learning for Drug Binding Structure Prediction. Arxiv,2022/6/4
相關文章:
1. Research and Markets發布新藥開發之AI應用市場現況與預測報告
2. 利用生物發光機制來開發新藥
3. 特斯拉將助CureVac打造「RNA 微型工廠」生產COVID-19疫苗
4. 美國國家衛生院授權WHO及MPP 11項COVID-19相關技術
5. 下一代COVID-19疫苗多樣化 可口服或噴劑
6. 淺談人類冠狀病毒疫苗開發策略
--------------------------------------------------------------------------------------------------------------------------------------------
【聲明】
1.科技產業資訊室刊載此文不代表同意其說法或描述,僅為提供更多訊息,也不構成任何投資建議。
2.著作權所有,非經本網站書面授權同意不得將本文以任何形式修改、複製、儲存、傳播或轉載,本中心保留一切法律追訴權利。
|